酶的高立体选择性和高活性是其应用于绿色生物制造的关键。然而,传统的酶工程方法(如定向进化)虽然有效,但通常需要消耗大量的实验资源和时间成本。近年来,计算预测工具的兴起为酶的理性设计提供了新的可能性,但现有工具在酶的精准设计方面表现受限。
近期,浙江工业大学生物工程学院郑裕国院士团队柳志强教授课题组开发了一种名为UniESA的数据驱动框架,旨在通过机器学习模型精准预测酶的立体选择性和活性。该框架基于氨基酸指数(AAindex)或蛋白质语言模型(PLM)编码,结合信号处理和机器学习回归算法,能够高效构建酶序列与立体选择性/活性之间的预测模型。UniESA的工作流程分为三个阶段:编码、建模和预测。首先,酶序列通过AAindex或PLM进行编码,生成特征向量。随后,利用多种机器学习回归算法对编码后的数据进行建模,并通过验证集评估模型性能。最后,选择表现最佳的模型用于预测高立体选择性和活性的酶突变体,并通过湿实验验证其性能。
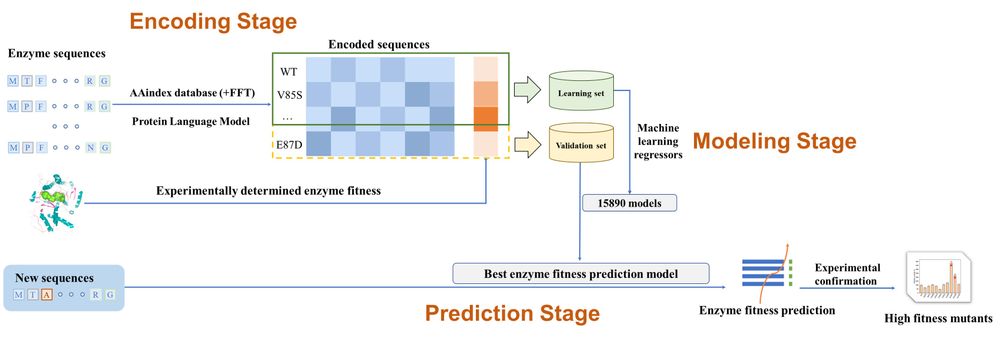
图1 UniESA的工作流程
多个公共数据集上的测试表明,UniESA在立体选择性和活性预测任务中表现出色(R2值0.65~0.98),并具有优良的普适性。

图2 UniESA在不同数据集中均表现优良
在羰基还原酶的进化任务中,通过对6,787个碳基还原酶(KmCR)单点突变的虚拟筛选,UniESA成功预测出两个具有严格立体选择性的突变体(dep>99.5%)和一个活性提高2.8倍的突变体,仅需传统定向进化实验工作量的十分之一甚至千分之一。这一突破为酶工程领域提供了一种高效、精准的理性设计工具,有望在绿色生物制造中大幅减少实验成本和时间。
相关研究成果以“UniESA: a unified data-driven framework for enzyme stereoselectivity and activity prediction”为题,于2025年3月发表于期刊Green Chemistry。我校柳志强教授为论文通讯作者,翁春跃副教授为论文第一作者。
浙江工业大学生物工程学院郑裕国院士团队柳志强教授课题组已在立体选择性羰基还原酶的筛选挖掘、催化机制解析、分子改造及手性醇的高效生物合成领域取得了一系列研究成果,实现了他汀类药物、度洛西汀、维贝隆、氟苯尼考等手性醇医药、农药中间体的绿色生物制造。
论文信息:Weng CY, Li J, Chen QL, et al. UniESA: a unified data-driven framework for enzyme stereoselectivity and activity prediction [J]. Green Chemistry, 2025。
论文链接:https://doi.org/10.1039/D4GC06253A

